Research Article
국내 작물의 배양 가능한 미생물 확인 및 유용 미생물 분리 방법 연구
장은지
1,†, 김라영
1,†, 김호연
2
, 이보경
1,*
Investigation of Total Culturable Bacterial Abundance and Isolation of Beneficial Bacteria from Agricultural Crops in South Korea
Eun Ji Jang
1,†, La Yeong Kim
1,†, Ho-Youn Kim
2
, Bokyung Lee
1,*
2한국과학기술연구원 천연물연구소 스마트팜융합연구센터
1Department of Food and Nutrition, College of Health Science, Dong-A University, Busan 49315, Korea
2Smart Farm Research Center, Korea Institute of Science and Technology (KIST), Gangwon 25451, Korea
*Corresponding author : Bokyung Lee, Department of Food and Nutrition, College of Health Science, Dong-A University, 840 Busan 49315, Korea. Tel: +82-51-200-7320, E-mail:
bolee@dau.ac.kr
† Both authors contributed equally to this study.
Copyright © Korean Society for Lactic Acid Bacteria and Probiotics. This is an Open-Access article distributed under the terms of the
Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/4.0/) which permits
unrestricted non-commercial use, distribution, and reproduction in any
medium, provided the original work is properly cited.
Received: Jun 08, 2020; Revised: Jun 25, 2020; Accepted: Jun 27, 2020
Published Online: Jun 30, 2020
Abstract
In this study, we investigated culturable bacterial abundance in various crops. Total culturable aerobic bacteria in tomato plants were enumerated on NA and it showed that the amounts of viable bacterial cells were changed among the different parts of the plants (roots, stems, and fruits), ranging between log 1 and 4 per g sample. To investigate the bacterial abundance dynamics in different crops, culturable aerobic bacterial cell numbers were enumerated on NA and compared between the samples (tomato fruit, sesame leaf, and green onion) harvested freshly from one field trial (Yangsan, Korea). In this trial, the number of total culturable aerobic bacteria of the samples were 8 times higher in sesame leaves and green onions compared to the tomato fruits in the field. Interestingly, culturable aerobic bacteria on MRS were only found in sesame leaves, whereas lactic acid bacteria were not detected in tomato fruit and green onion samples in this field. We also found that the field location (Yangsan, Gangneung, or Busan) influenced the number of culturable aerobic bacteria on the surface of the crops. Tomato samples freshly harvested from the different field locations were tested and showed that the amounts of culturable aerobic bacteria on NA and MRS agar were significantly different based on the field locations of the samples. Finally, lactic acid bacteria were isolated from all the tomato and sesame leaf samples used in this study, showing 20 and 28 morphological diversity. With these isolates, we will be able to conduct further biological and functional investigation to develop a new probiotic strain originated from the crops in Korea.
Keywords: lactic acid bacteria; sesame leaf; tomato; potential probiotic strains
서론
2002년 세계보건기구(WHO)와 국제식량농업기구(FAO)에서는 ‘프로바이오틱스란 유효한 수준을 섭취하였을 때 숙주의 건강증진을 가져오는 살아있는 미생물’이라 정의하였다(WHO/FAO, 2002). 대부분의 프로바이오틱 균주들은 유산균에 속하며, 숙주 내 유해 균의 증식을 저해하는 방식으로 숙주의 건강 증진에 이바지한다(Ahn, 2011). 현재 다양한 프로바이오틱스 제품이 질병(변비, 설사, 장염 및 혈중 콜레스테롤 등)을 예방하고 치료하는 목적으로 소비되고 있으며, 그 시장이 지속적으로 확대되고 있다(Ahn, 2011). 프로바이오틱스의 주요 미생물인 유산균은 유제품, 김치, 젓갈 등의 발효식품에 풍부하게 존재하며(shin et al., 2017), 프로바이오틱 미생물로 알려진 유산균들은 대부분 Lactobacillus spp. 및 Bifidobacterium spp.에 속하며(Ahn, 2011), 김치의 주요 발효 유산균으로는 Leuconostoc, Lactobacillus, Weissella와 Pediococcus spp. 등이 있다(Lee et al., 2016).
유산균은 발효식품뿐만 아니라, 재배되고 있는 다양한 작물 표면 등 천연물에도 서식하며, 자연계에 넓게 분포되어 있다(Park, 2019). 미생물은 다양하고 복합적인 환경에서 존재하기 때문에(Johnson et al., 2015), 미생물 군집을 이해 및 예측하고 통제하는 것은 다양한 연구에서 잠재적인 응용 분야의 핵심으로 작용될 수 있다(Wolfe et al., 2015). 따라서 본 연구에서는 국내 다양한 지역의 토양에서 키운 작물을 이용하여 유산균을 추출하되, 다른 지역에서 자란 같은 종류의 작물의 균 수와, 같은 지역에서 자란 다른 종류의 작물들의 균수를 중점적으로 비교하였다. 이를 통해 다양한 재배지역과 작물에 따른 유산균의 분리에 대해 그 실험방법을 연구하고자 하였다. 또한, 주변에 존재하는 다양한 채소 및 천연물질로부터 유산균을 분리하여 분포를 조사하고, 다양한 작물 유래의 잠재적 프로바이오틱스 후보 유산균 확보하고자 하였다.
재료 및 방법
시약 및 재료
본 연구에서는 토마토와 깻잎, 대파가 시료로 사용되었으며, 토마토는 한국과학기술연구원 천연물연구소 스마트팜융합연구센터와 거제 시장의 대저 원산지의 토마토, 양산에 있는 텃밭에서 제공받았고, 각각 강릉, 대저, 양산으로 명명하여 진행했다. 실험은 총 4번으로 이루어졌으며 첫 실험에서는 한국과학기술연구원에서 제공받은 토마토 전초를 이용하여 부위별(과실, 가지, 줄기, 뿌리)로 표면 미생물의 분포를 알아보았고, 두 번째 실험에서는 같은 토마토 전초의 과실만을 사용하여 크기별로 표면 미생물의 잠재적인 총 균 수와 유산균을 검출하기 위해 1.5%의 agar(BD, sparks, MD, USA)가 포함된 Nutrient broth(BD, sparks, MD, USA, 이하 NA agar)와 MRS(BD, sparks, MD, USA, 이하 MRS agar)에 배양하였다. 세 번째 실험에서는 대저 토마토를 이용하여 미생물을 분리했으며, 마지막 실험은 양산 토마토, 깻잎의 미생물 분포를 알아보았다. 미생물 분리에 사용한 것은 0.1% peptone buffer(BD, sparks, MD, USA), sonicator(Brason, 2510EDTH, 40kHz, USA), 원심분리기(gyrozen, LaboGene 1580R, Korea)이며, 잠재적인 총균수를 알아보기 위해 NA agar를 사용하였다. 또한 잠재적인 유산균의 분리를 위해 MRS agar를 사용하였다. 실험 결과는 GraphPad prism 8.4.3(GraphPad SoftWare Inc., USA)을 이용하여 그래프로 나타내고 통계분석을 진행하였다. 한 그래프 안의 두 개의 시료 비교 시 unpaired Student’s t-test를, 세 개의 시료를 비교 시 One-way ANOVA를 사용하였다.
토마토 전초의 부위별 미생물 분리
토마토 전초의 표면 미생물을 생균수 측정을 위하여 8가지 샘플(과실(162.5 g), 과실(19.9 g), 가지1, 가지2, 줄기, 뿌리, 잎, 흙)을 준비하였다. 부위별로 구분한 토마토 전초의 표면 미생물을 분리하기 위해 먼저 시료의 무게를 측정한 후 시료를 멸균된 0.1% peptone(BD, sparks, MD, USA) buffer에 넣어 ultra sonication을 10분 진행하였다(Thomas et al., 2013). 이의 총균수를 관찰하기 위해 NA agar에 ultra sonication이 끝난 샘플용액(0.1% peptone buffer)을 0.5 mL씩 spreading하여 30℃에서 24시간 배양하였다.
토마토 전초의 원심분리 전후 총 균수 차이 확인
앞서 10분간 ultra sonication한 0.1% peptone 용액을 원심분리(3,220 ×g, 15분, 4℃)한 후(Thomas et al., 2013), 이 중 상등액의 생균수 측정을 위하여 NA agar에 0.5 mL씩 spreading하여 30℃에서 24시간 배양하였다. 이는 원심분리 전후 총균수 차이를 비교하여 원심분리가 잘 이루어졌는지를 살펴보기 위해 실행하였다. 원심분리 후, pellet은 후속연구에서 마이크로바이옴 분석을 위하여 –80℃에 보관하였다.
토마토 과실의 표면 미생물 분리
토마토 전초에서 크기에 상관없이 임의로 과실 6개를 선정하여 추가적으로 미생물 분리를 하였다. 토마토는 큰 과실, 중간 과실, 작은 과실의 3분류로 구분하여 크기별 2개씩 총 6개의 과실을 임의로 선정하고, 시료의 무게를 측정한 후 멸균된 0.1% peptone 용액을 과실이 잠길 정도로 각각 넣어 큰 과실 20분, 중간 과실 15분, 작은 과실은 10분으로 ultra sonication을 실시하였다(Thomas et al., 2013). 총균수를 알아보기 위해서 NA agar에 1 mL씩 spreading하였고, 잠재적인 유산균을 분리하기 위하여 MRS agar에 1 mL spreading하여 30℃에서 24시간 배양하였다. 추후 마이크로바이옴의 분석을 위해 원심분리(3,220 ×g, 15분, 4℃)하여 상등액은 폐기하고 pellet만 –80℃에 보관하였다.
토마토, 깻잎, 대파의 표면 미생물 분리
각 시료의 무게를 측정한 후, 멸균된 0.1% peptone buffer를 시료가 잠길 만큼 넣어 20분간 ultra sonication을 실시한 후 미생물을 분리하였다. 총균수와 유산균을 조사하기 위해 Ultra sonication이 완료된 0.1% peptone의 원액과 10배 희석한 용액을 NA agar와 MRS agar에 1 mL씩 spreading하여 30℃에서 24시간 배양하였다. 또한, 원액을 원심분리(3,220 ×g, 15분, 4℃)하여 pellet만 –80℃에 보관하여 이후 작물 마이크로바이옴 패턴을 분석하고자 하였다. 양산 토마토와 깻잎, 대파의 미생물 분리는 두 차례에 걸쳐 반복하여 진행하였다.
시료별 분리한 균의 수 계산
시료별로 표면 미생물을 분리하여 NA 한천배지와 MRS 한천배지에 자란 균을 {균수×사용한 0.1% peptone(mL)}/{시료의 무게(g) ×A}×희석배수(A: spreading 시 분주한 0.1% peptone buffer)의 식을 이용하여 log(CFU/g)의 값으로 표현하였다.
MRS 한천배지로부터 얻은 균 분리
앞서 각 시료별로 MRS agar로부터 얻은 균을 단일 colony를 얻기 위해 streaking한 후, 형태학적으로 구분하였다. 단일 colony를 MRS broth에 접종하여 30℃에 24시간 배양하고, 균이 자라면 25% glycerin stock을 제조하여 –80℃에 보관하였다.
결과 및 고찰
토마토 전초의 다양한 부위에서 배양 가능한 총균수 확인
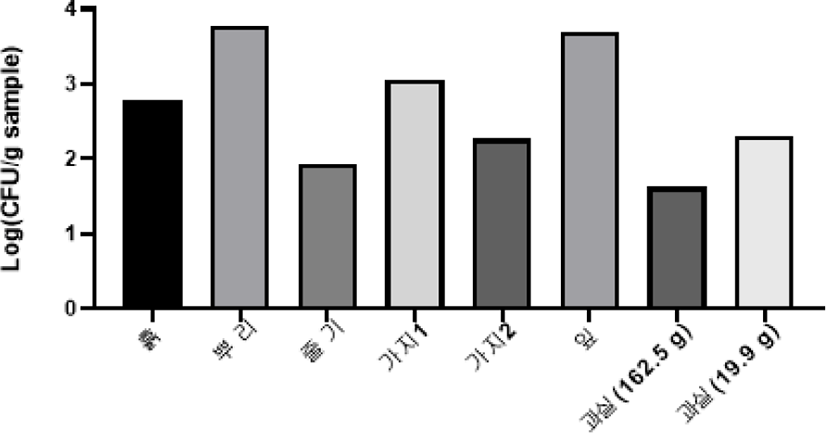
Fig. 1은 토마토 전초로부터 분리한 미생물을 NA agar(이하 NA 한천배지)에서 배양가능한 균을 관찰한 결과이다. 토마토 전초를 과실 2개(162.5 g, 19.9 g), 뿌리, 줄기 가지1, 가지2, 잎으로 구분하였으며, 부가적으로 흙의 잠재적인 총균수를 파악하고자 하였다. 8개의 시료로부터 NA 한천 배지를 통해 얻은 균은 시료 1 g당 약 474.5 CFU이 검출되었으며, 특히 뿌리에서 5,888.4 CFU/g으로 가장 많은 균이 존재하고 있음을 확인하였다. 이는 약 41.7 CFU/g으로 가장 적은 양의 균이 검출된 과실(162.5 g)의 균수와 비교했을 때, 약 141배 많은 균이 존재한다는 것을 확인하였다. Seo(2017)에 따르면 뿌리 채소인 우엉, 연근, 칡의 추출물 모두에서, 5종의 공시 유산균이 107 CFU/mL 이상으로 생육하는 것이 확인되었다. 이를 통해, 뿌리 식물 또는 식물의 뿌리에 존재하는 유산균을 비롯한 미생물의 분리 연구가 필요할 것이라 판단된다. 토마토 과실의 경우, 약 4.8 CFU/g의 차이로 과실(19.9 g)이 더 많은 양의 균이 검출되었으므로 토마토 과실에도 균이 존재함을 확인하였다.
Fig. 1.
Total culturable bacterial abundance on the surface of tomato plants. Total culturable aerobic bacteria of tomato plants were enumerated on NA agar. Samples (tomato roots, tomato stems, tomato leaves, fruits and soil) were freshly collected from Smart Farm Research Center in Gangneung.
Download Original Figure
토마토 과실에 존재하는 배양 가능한 미생물의 지역별 차이
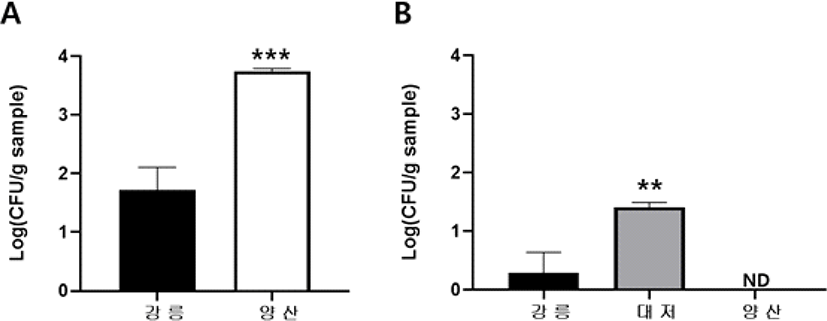
앞선 실험을 통해 토마토 과실에도 균이 존재한다는 것을 파악한 후, 다른 지역의 토마토 과실의 균의 존재 유무를 조사하기 위해 두 지역(부산광역시, 경상남도)을 선정하였다. Fig. 2는 부산광역시와 경상남도에서 재배된 토마토 과실에 존재하는 균을 NA 한천 배지와 MRS agar(이하 MRS 한천 배지)에 spreading하여 분리한 균수를 나타낸 것이다. 강릉 토마토와 양산 토마토를 NA 한천 배지에 배양한 결과, 각각 약 5,011.9 CFU/g, 약 50.1 CFU/g의 균이 검출되었으며, 약 100배 이상 차이가 난다는 것을 확인하였다(Fig. 2A). 해당 경우 양산 토마토는 99.9%의 신뢰구간 안에서 통계적으로 유의적인 차이가 난다는 것을 확인하였다. 잠재적인 유산균을 검출하고자 MRS 한천 배지에 spreading하여 배양한 균은 세 지역 중 대저에서 25.9 CFU/g으로 가장 많이 검출되었으며, 99% 신뢰구간 안에서 유의적으로 차이가 났다. 또한, 강릉 토마토의 검출량인 1.9 CFU/g의 25.12배만큼의 균이 검출되어 잠재적 유산균은 세 지역 중 대저가 가장 많은 것으로 파악할 수 있다(Fig. 2B). 양산 토마토에서는 균이 검출되지 않았다(Fig. 2B). 양산 토마토는 잠재적인 총균수는 많지만, 다른 지역에 비해 잠재적 유산균이 존재하지 않는 것으로 결론 내렸다(Fig. 2A, Fig. 2B). Kim (2018)에 따르면 시료의 채취 시기 및 지역별 microbiota 군집 분석을 한 결과, 몇몇 샘플은 채취 지역 간의 명확한 차이가 발견된 것을 확인할 수 있었다. 이를 통해 같은 종류의 샘플이라도 배양된 지역에 따라 균수 및 종류의 차이가 나타나는 것으로 판단된다. 농림식품기술기획평가원의 ‘토마토 추출물을 포함하는 유청 배지 제조 및 유산균의 배양’ 기술에 따르면 토마토의 추출물로 인해 미생물 배양용 배지를 제조하는 것이 가능한 것으로 나타나 있어 토마토에도 유산균이 존재함을 뒷받침하였다.
Fig. 2.
Culturable surface bacterial cell numbers of tomato fruits from different fields. Culturable aerobic bacteria of tomato fruits were enumerated on NA(A) or MRS(B) agar. Tomato fruits were grown in different locations (Gangneung or Yangsan) and freshly harvested for this study (A, B) and Daejeo tomato fruits were from farmer’s market in Busan (B). The average ± std of two to three individual fruits is shown for each bar (***:p<0.001 by unpaired student’s T test(A); **:p<0.01 by one-way ANOVA(B), ND: not detected).
Download Original Figure
양산 작물의 잠재적 총 균수와 유산균수가 작물에 따라 다르다.
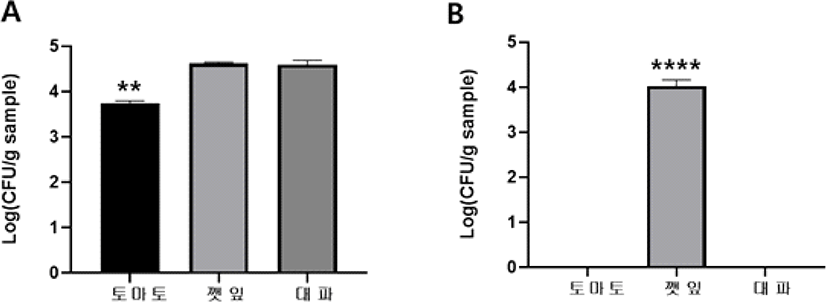
앞서 토마토 전초를 부위별로 구분하여 NA 한천 배지에 배양함으로써, 표면 미생물의 생균을 관찰하여 뿌리와 잎, 가지에서 많은 양이 검출됨을 확인하였다. 이에 잠재적인 총균수와 유산균수에 대하여, 같은 지역 내의 다양한 작물에 따른 차이를 알아보고자 토마토 전초의 가지, 잎과 비슷한 작물인 대파와 깻잎, 그리고 토마토를 통해 잠재적인 총균수와 유산균수를 알아보았다(Fig. 3). 작물의 표면 미생물을 분리하여 NA 한천 배지에 배양하여 얻은 균은 가장 적은 양이 검출된 토마토와 비교했을 때, 깻잎에서 1 g당 약 7.9배 이상이 검출되었다(Fig. 3A). Fig. 3A의 시료를 비교했을 때 깻잎은 약 log 4.6 CFU/g, 대파는 약 log 4.5 CFU/g으로 큰 차이가 보이지 않았으며, 토마토는 99%의 신뢰구간 안에서 유의적인 차이를 보였다는 결과를 도출했다. Fig. 3B의 경우, 잠재적인 유산균 수를 확인했을 때 토마토와 대파에서는 검출되지 않았지만 깻잎에서 약 10,000배 이상의 균이 검출되었음을 알 수 있다. Fig. 2B의 대저 토마토와 Fig. 3B의 깻잎을 비교했을 때 깻잎이 대저 토마토에 비해 100배 이상의 잠재적인 유산균 수를 갖고 있는 작물이라는 점도 파악하였다. Hong(2013)에 따르면 BCP 지시약이 첨가된 MRS agar plate에 100μL씩 spreading하여 37℃에서 48시간 동안 배양하였을 때, 채소류에서 210주, 청국장에서 110주, 메주에서 58주를 분리하였다. 이를 통해 발효식품뿐만 아니라, 채소류에서도 다량의 유산균이 검출됨을 알 수 있다.
Fig. 3.
Total culturable aerobic bacteria on tomato fruit, sesame leaf, and green onion in the same field. Culturable surface bacteria of different crops from the same filed were enumerated on NA(A) or MRS(B) agar. The average ± STD of two to three individual crops is shown for each bar (**: p<0.01, ****: p<0.0001 by One-way ANOVA). ND: not detected.
Download Original Figure
형태학적 특성에 따른 MRS 한천배지에서 자란 균 구분
Table 1은 각 시료에서 분리한 미생물을 MRS 한천배지에서 30℃, 24시간 배양하여 얻은 균을 형태학적으로 구분한 그래프이다. 형태학적으로 토마토 과실은 20종류의 균이 검출되었으며, 깻잎은 28종류의 균이 검출되었다. 대파의 경우, MRS 한천배지에서 균이 검출되지 않았다. Ko et al.(2013)에 따르면 5개 지역의 다양한 종류의 김치로부터 유산균을 분리하였는데, 그 결과 124종의 갓김치, 62종의 열무김치, 99종의 배추김치, 33종의 깍두기로부터 각각 129종, 53종, 122종, 44종의 유산균을 분리하였다. 이와 비교했을 때 토마토 과실과 깻잎은 각각 한 가지 종으로부터 20종 이상의 유산균을 분리하였으며, 이는 토마토 과실과 깻잎 표면에 보다 다양한 종의 유산균이 존재하는 것으로 판단할 수 있다.
Table 1.
Morphological classification of lactic acid bacteria from freshly collected crops
|
작물 종류 |
균주 수(형태학적 (morphology) 특성에 따른 구분 |
|
토마토 |
20 |
|
깻잎 |
28 |
|
대파 |
NA |
Download Excel Table
원심분리와 ultra sonication 시간의 적절성
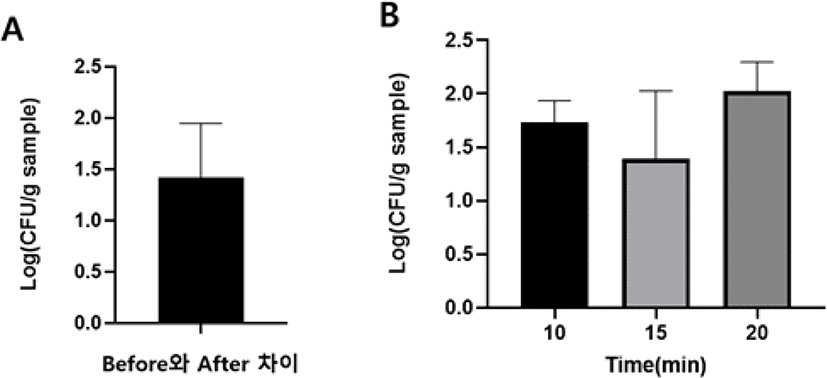
Fig. 4는 적절한 원심분리방법과 ultra sonication시간을 도출하고자 실험에서 사용한 원심분리방법과 ultra sonication 시간을 균 log(CFU/g)과 비교한 것이다. 토마토 전초 실험에서 사용한 원심분리(3,220 ×g, 15분, 4℃) 방법을 통해 원심분리 전후의 균수 차이를 관찰함으로써 적절한 원심분리 조건인지 판단했다. 원심분리 이후 NA 한천배지를 통해 검출한 균이 원심분리 이전에 비해 96% 이상이 제거됨을 확인하였으며, pellet에 정상적으로 미생물이 축적되었기 때문에 마이크로바이옴 패턴 분석 시 문제가 없고, 해당 원심분리 방법이 적절하다는 결론을 내렸다(Fig. 4A). Fig. 4B에서는 시료 당 실시한 ultra sonication 시간에 따른 시료들의 균 log(CFU/g)의 평균을 나타낸 것으로, 가장 적은 균이 검출된 시간인 15분보다 가장 많은 균이 검출된 시간인 20분에서 약 4배 더 검출되었다. 차이를 확인할 수 있지만 통계적인 차이는 없음을 확인하였다. 20분간 ultra sonication시 분리된 생균은 약 105.7 CFU/g의 양상을 띄므로 가장 적절한 것으로 판단했다.
Fig. 4.
Optimization of sample preparation to enumerate the surface bacterial population from freshly collected crops. Culturable bacteria was enumerated before and after centrifugation (A) and from the samples processed in different sonication time (minutes) (B).
Download Original Figure
작물 차이에 따른 미생물 분리 시 사용할 1 g당 0.1% peptone(mL) 도출
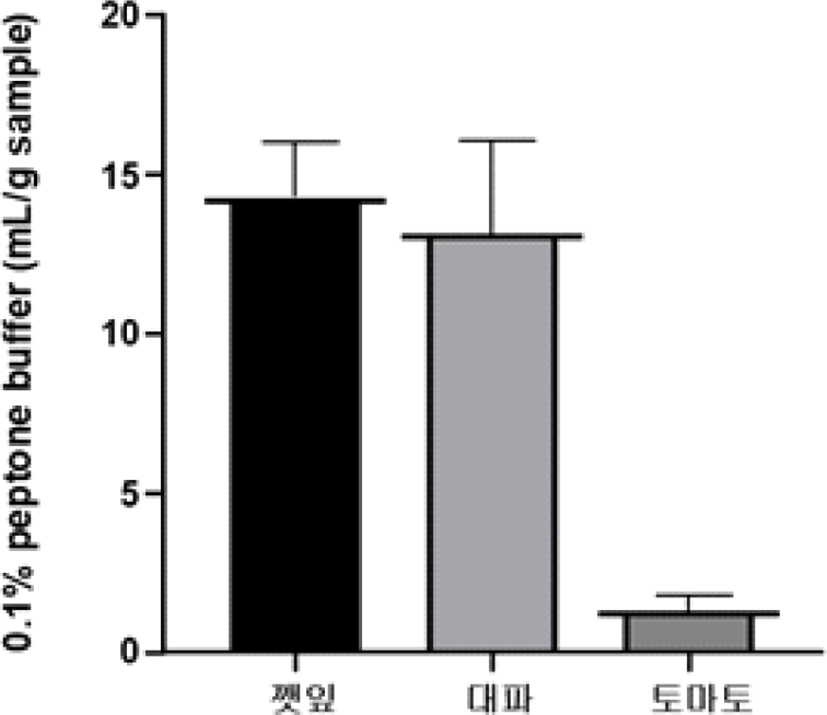
Fig. 5는 다양한 작물(토마토, 깻잎, 대파)을 이용한 실험 당시, 미생물 분리를 위해 사용했던 시료 1 g당 0.1% peptone buffer의 평균을 나타낸 것이다. 깻잎은 1 g당 약 14.3 mL, 대파는 1 g당 약 13.1 mL가 사용되었으며, 토마토는 1 g당 약 1.3 mL가 사용되었다. 대파와 깻잎은 토마토 과실에 비해 약 10배 정도의 0.1% peptone buffer가 필요하며, 시료 1 g당 10 mL 이상의 0.1% peptone buffer가 필요한 것으로 판단하였다. 토마토는 99.9% 신뢰구간 안에서 유의적으로 차이가 있으며, 1 g당 약 1.5 mL 이상의 0.1% peptone buffer가 적절한 양이라는 결론을 내렸다.
Fig. 5.
Volume of 0.1 % peptone buffer (mL) per g sample used for sonication to dislodge the bacterial cells.
Download Original Figure
감사의 글
본 연구는 동아대학교 2019년도 신임교원과제 연구비로 수행되었음.
References
Ahn Y (2011) [Lactic Acid Bacteria] Probiotic lactic acid bacteria.
Korean J. Food & Nutr.24(4), 817-832.


Yoon JA and Shin K (2017), Studies on the function of lactic acid bacteria and related yeasts in probiotics: A review,
Korean J. Food & Nutr.30(3), 395-404.

Lee X, Lee HA, Kweon M, Park E, and Park K (2016) Probiotic effects of
Lactobacillus plantarum strains isolated from Kimchi.
Korean J. Food & Nutr.45(12), 1717-1724.


George F, Daniel C, Thomas M, Singer E, Guilbaud A, Tessier FJ, Revol-Junelles A, Borges F, and Foligné B (2018) Occurrence and dynamism of lactic acid bacteria in distinct ecological niches: A multifaceted functional health perspective.
Front Microbiol.
9, 2899. doi:




Johnson DR, Helbling DE, Lee TK, Park J, Fenner K, and Kohler H-PE
et al. (2015) Association of biodiversity with the rates of micropollutant biotransformations among full-scale wastewater treatment plant communities.
Appl Environ Microb.81, 666-675.




Wolfe BE and Dutton RJ (2015) Fermented foods as experimentally tractable microbial ecosystems,
Cell.161(1), 49-55.



Williams TR, Moyne AL, Harris LJ and Marco ML (2013) Season, irrigation, leaf age, and
Escherichia coli inoculation influence the bacterial diversity in the lettuce phyllosphere.
PLoS One.8, e68642.




Kim TY, Lee J, Kim B, and Choi SH (2017) Whole-body microbiota of sea cucumber (
Apostichopus japonicus) from South Korea for Improved Seafood Management.
J. Microbiol. Biotechnol.27(10), 1753-1762.



Yoon S
et al. (Office of Research Affairs UIF, YONSEI UNIVERSITY). Manufacture of
Lactobacillus and culture of
Lactobacillus containing tomato extracts, patent number 1016141450000.

Hong SW, Bae HJ, Chang JH, Kim SY, Choi EY, Park BY, Chung KS, and Oh MH (2013) Isolation and identification of bacteriocin-producing lactic acid bacteria.
Korean J. Dairy Sci. Technol.31(2), 153-159.

Ko KH, Liu W, Lee HH, Yin J and Kim IC (2013) Biological and functional characteristics of lactic acid bacteria in different Kimchi.
Korean J. Food & Nutr.42(1), 89-95.