서 론
최근 전세계적인 COVID-19의 판데믹 유행에 따라 건강에 대한 국민적 관심사가 급증하면서 건강 기능식품 시장은 급격한 성장을 기록하고 있다. 그 중에서도 유산균 프로바이오틱스 시장은 2022년 시장규모 1조원을 넘길 것으로 예상되고 있으며, 홍삼에 이어 두번째로 큰 시장규모를 가질 것으로 예상된다. 이렇게 시장이 확대됨에 따라 소비자들의 유산균에 대한 관심도 급증하고 있고, 제조사에서도 유산균 제품에 대한 안전성 연구를 강화하고 있다. 그러나 생균을 직접 사용하는 프로바이오틱스 제품의 특성상, 고용량으로 투여된 균은 사용자의 장내미생물 균총 및 신체 건강에 직접적인 영향을 줄 수 있기에 긍정적 효능과 더불어 일부의 부작용도 우려되고 있다. 실제 유산균의 사용에 따른 감염으로 인해 사망한 사례들이 보고된 적이 있고[1], 국내의 경우 건강기능 식품 이상 사례 신고센터에 접수된 누적사례 중 유산균과 관련된 사례가 1,300여건이 보고되기도 하였다(https://foodsafetykorea.go.kr/minwon/sideeffect/stats.do). 이런 다량의 생균의 사용은 또한 항생제 다제내성균의 발생에도 영향을 줄 수 있는데 특정 세균의 경우 수평적 유전자 이동이라는 메커니즘을 통해 항생제 저항성 유전자가 다른 미생물로 전파될 가능성이 있기 때문이다[2]. 이에 따라 안전한 프로바이오틱스 제품의 개발을 위해서는 사용되는 유산균의 항생제 저항성 유무를 확인하는 것이 필수적이다. 특히 유산균의 유전적 특성, 그 중에서도 항생제 저항성 유전자를 정확하게 파악해야 한다. 이런 이유로 2018년 식품의약품안전평가원에서는 고시형 프로바이오틱스 19종에 포함됨에도 항생제 저항성 관련 이슈가 있는 Enterococcus 2종 (E. faecalis, E. faecium)에 대해 유전적 특성 검증 후 제품에 사용하는 허가제로 변경되었다. 이에 따라 실제 항생제 투여실험을 통해 항생제 저항성과 관련된 유산균의 표현형적 특성을 파악할 수 있는 실험을 수행해야 한다. 아울러 생물정보학적 분석을 통해 유전체 내에 존재하는 항생제 저항성 유전자를 탐색하는 연구도 필요하다. 한편, 항생제 저항성 실험과 관련된 기법들은 일정 정도 표준화되어 있는 것에 반해 유전체 내에 포함된 항생제 저항성 유전자의 검출과 관련된 유전체 분석의 경우 그렇지 않은 문제점이 있다. 생물정보학적 분석방법이 매우 다양할 뿐만 아니라, 분석방법에 따라 결과 또한 많은 차이를 보여 해당 유산균이 가지고 있는 잠재적인 위험성을 객관적으로 파악하는데 어려움이 있다. 이런 문제점을 해결하기 위해 본 연구에서는 유산균의 유전체에 포함되어 있는 항생제 저항성 유전자의 분석 방법에 따른 검출 결과의 차이와 경향성을 비교해 보았다. 이번 연구 결과는 분석 플랫폼의 표준화와 항생제 저항성 유전자 분석기준에 대한 기본 자료로 활용될 수 있을 것으로 기대한다.
재료 및 방법
건강기능식품 고시형 프로바이오틱스 유산균 19종에 해당하는 유산균들의 유전체를 수집하였다. NCBI Refseq Database[3]를 기준으로 공개된 유전체 중에서 Assembly Level이 Complete인 유전체만을 모두 다운로드하여 분석을 수행하였다. 사용된 유전체의 수는 종별로 Lactobacillus acidophilus 8개, Lacticaseibacillus casei 3개, Lacticaseibacillus paracasei 34개, Lactobacillus delbrueckii 2개, Limosilactobacillus fermentum 27개, Lactobacillus gasseri 9개, Lactobacillus helveticus 15개, Lactiplantibacillus plantarum 142개, Limosilactobacillus reuteri 24개, Lacticaseibacillus rhamnosus 26개, Ligilactobacillus salivarius 11개, Bifidobacterium animalis 8개, Bifidobacterium bifidum 9개, Bifidobacterium breve 37개, Bifidobacterium longum 24개, Streptococcus thermophilus 68개, Lactococcus lactis 13개, Entetococcus faecalis 87개, Enterococcus faecium 235개로 총 782개의 유전체가 본 연구에 사용되었다.
항생제 저항성 유전자를 분석을 수행할 수 있는 분석 플랫폼과 데이터의 형태는 현재 여러가지 종류가 활용이 가능하다. 그러나, 조립이 되지 않은 Shotgun Sequencing 데이터의 경우 유전체가 절편화 되어 유산균의 완전한 유전자의 파악이 제한적이고, 유산균 사용 전 허가의 기준이 Complete Genome인 점을 감안하여 Assembled Genome을 대상으로 사용할 수 있는 분석 플랫폼을 기준으로 본 연구의 분석 tool로 적용하였다. 그 중 다수의 분석 수를 감안하여 Web상이 아닌 Local에서 Standalone으로 수행할 수 있는 프로그램 중 주요 항생제 저항성 유전자 데이터베이스에서 배포하는 대표 분석 tool들을 위주로 선발하였으며, 대표 tool이 배포되지 않는 데이터베이스의 사용을 위해 여러 항생제 저항성 유전자 데이터베이스를 종합적으로 사용할 수 있는 ABRicate를 동시에 사용하였다. 최종적으로 본 연구에 사용된 분석 플랫폼은 총 4개로, rgi (Resistance Gene Identifier: CARD AMR Database)[4], AMRFinderPlus (NCBI AMR Database) [5], staramr (resfinder AMR Database)[6, 7], ABRicate (Multi AMR database)[8-12]이다. 분석 결과의 시각화는 pheatmap R package[13]를 이용하였다.
결 과
Fig. 1은 종별로 사용된 전체 유전체 중, 4개의 분석 플랫폼에서 항생제 저항성 유전자가 검출된 유전체의 비율을 Heatmap으로 보여준다. Enterococcus 속(genus)과 Limosilactobacillus fermentum, Limosilactobacillus reuteri, Bifidobacterium animalis를 제외한 대부분의 건강기능식품 고시형 유산균들은 사용되는 분석 플랫폼에 따라 항생제 저항성 유전자의 검출결과가 차이가 나는 것을 확인할 수 있다. 사용된 모든 플랫폼에서 거의 동일한 결과를 내는 종들도 일부 있었으나, 대부분의 경우 각 플랫폼별로 항생제 저항성 유전자가 검출되는 유전체가 달랐고 대표적인 항생제 저항성 유전자 데이터베이스인 CARD database를 이용하는 분석 tool인 rgi의 경우 상대적으로 다른 분석 tool에서는 검출되지 않는 항생제 저항성 유전자가 단독으로 검출되는 경향을 보였다. 그림상에서 rgi에서도 항생제 저항성 유전자가 검출되지 않은 것으로 보이는 Limosilactobacillus fermentum의 경우에도 총 27개의 유전체 중 2개의 유전체에서 rgi tool에서만 항생제 저항성 유전자가 검출되었다(major facilitator superfamily (MFS) antibiotic efflux pump, CMY beta-lactamase). 고시형 19종으로 사용되는 유산균 중에서 높은 항생제 저항성 유전자 전파 가능성을 가지고 있어 건강기능식품으로의 사용이 엄격해지는 Enterococcus 2종 (E. faecalis, E. faecium)의 경우 분석 tool에 상관없이 사용된 322개의 유전체 모두 항생제 저항성 유전자를 가지고 있는 것으로 확인되었다. 분석 tool별 경향성을 살펴보면 AMRFinderPlus와 ABRicate가 가장 유사한 양상을 보였다. staramr 플랫폼도 rgi의 결과에 비해서는 두 분석 tool과 상대적으로 유사하였으나 일부 종의 분석에서는 검출 결과에 차이를 보였다.
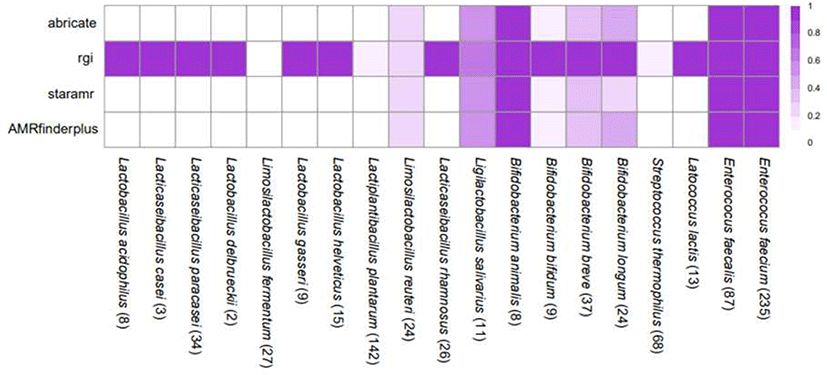
유전체 별 항생제 저항성 유전자의 단순 검출 유무 외에도 동일한 유전체 서열에 대해 검출되는 항생제 저항성 유전자의 종류에서도 사용되는 tool에 따라 차이를 보였다. Table 1은 11개의 Ligilactobacillus salivarius종 유전체에서 각 tool별로 검출되는 항생제 저항성 유전자를 보여준다. 총 발견된 12개의 항생제 저항성 유전자 중에서 9개의 항생제 저항성 유전자는 사용된 4개의 tool 모두에서 검출되었으나, 3개의 유전자 중 aminoglycoside nucleotidyltransferase(ANT(9))는 staramr에서는 검출되지 않았고 non-erm 23S ribosomal RNA methyltransferase와 copper-translocating P-type ATPase tcrB의 경우 각각 rgi tool과 AMRFinderPlus에서만 해당 항생제 저항성 유전자가 검출되었다. Bifidobacterium longum에서도 비슷한 양상을 보였는데 Table 2는 각 tool별로 Bifidobacterium longum에서 검출된 항생제 저항성 유전자를 보여준다. 4개의 tool에서 검출된 총 10개의 유전자 중에서 3개의 유전자만이 4개의 분석 tool 모두에서 검출되었고 2개의 유전자(aminoglycoside nucleotidyltransferase(ANT(6)), streptothricin acetyltransferase(SAT4))는 Ligilactobacillus salivarius 와 마찬가지로 staramr에서는 검출되지 않았으며 나머지 5개 유전자는 rgi tool에서만 검출되었다. 전체 검사결과를 종합해서 비교해 보았을때, rgi 분석 tool이 가장 많은 항생제 저항성 유전자를 검출하는 결과를 보였으며, 그 다음으로는 AMRFinderPlus가 많은 항생제 저항성 유전자를 검출하였다. staramr과 ABRicate는 유사한 검출 정도를 보였다. ABRicate를 제외한 나머지 3개의 tool이 단일 항생제 저항성 유전자 데이터베이스를 사용하는데 반해 ABRicate는 NCBI, CGE, ARG-ANNOT, CARD, MEGARes 등 다른 분석 tool이 사용하는 항생제 저항성 유전자 데이터베이스를 복합적으로 사용한다. 그럼에도 불구하고, 분석결과를 비교해 보았을 때, 단일 데이터베이스를 사용하는 tool의 검출 결과에 포함되는 항생제 저항성 유전자가 검출되지 않는 경우가 상당수 있음이 확인되었다.
고 찰
본 연구의 분석결과에서 확인할 수 있듯이 동일한 유전체 서열이라고 하더라도 항생제 저항성 유전자의 검출 분석에 사용되는 tool에 따라 그 결과가 매우 달라지는 것을 확인할 수 있다. 이렇게 분석 tool마다 결과가 다르게 나타나는 원인은 여러가지가 있을 수 있는데, 먼저 각 tool이 항생제 저항성 유전자를 판별하기 위해 사용하는 유전자 데이터베이스의 차이가 있을 수 있다. 본 연구에서 사용된 분석 tool 중 3개는 각기 고유한 항생제 저항성 유전자 데이터베이스를 사용하는데, 각 데이터베이스의 업데이트 정도와 데이터베이스별 특정 유전자의 항생제 저항성 유전자 포함 여부에 따라 검출결과가 달라질 것이 예상된다. 그러나 여러 종류의 데이터베이스를 복합적으로 사용하고 있는 ABRicate의 분석 결과가 각 단일 항생제 저항성 유전자 데이터베이스를 사용하는 대표 분석 프로그램과 다른 결과를 보이는 것과 데이터베이스에 해당 유전자가 존재함에도 특정 tool에서만 해당 유전자가 검출되는 결과를 보았을 때, 분석 결과의 차이는 사용되는 데이터베이스 차이 외에도 항생제 저항성 유전자가 있음을 판단하는 검출 알고리즘의 차이가 분석 tool 간의 다른 결과에 상당한 영향을 주는 것을 예상할 수 있다. 실제로 이런 차이로 인해 AMRFinderPlus(NCBI)에서는 ABRicate에서 사용하는 NCBI AMR 데이터베이스가 Full set이 아닌 Subset이며, 검출 알고리즘의 차이로 인해 두 tool의 분석 결과가 동일하게 나오지 않는 경우가 자주 발생하기 때문에 ABRicate가 AMRFinderPlus의 데이터베이스를 포함하여 사용함에도 AMR FinderPlus를 이용한 분석을 수행할 것을 권장하고 있다. 이처럼 각 tool별로 항생제 저항성 유전자의 유무 확인을 위해 사용하는 분석 알고리즘에 따라서도 동일한 서열에 대해 다른 결과가 나타나기 때문에 항생제 저항성 유전자 분석을 수행함에 있어서 주의가 필요할 것으로 생각된다.
2021년 7월, 식품의약품안전평가원에서는 건강기능식품 기능성 원료 중 프로바이오틱스에 사용하는 유산균의 안전성을 객관적으로 평가하는 가이드라인를 제시하였다(https://www.mfds.go.kr/brd/m_1060/view.do?seq=14866). 해당 가이드라인에는 안전성 평가 시험에 대해 총 4가지 실험적인 항목(항생제 내성, 용혈활성, 독소 생성, 대사적 특성)의 시험법과 더불어 항생제 저항성 유전자의 분석법을 제안하였다. 그 중 본 연구의 대상인 항생제 저항성 유전자의 경우 기존 고시형19종의 유산균 중에서 Enterococcus 속(genus) 2종에 대해 사용 전 항생제 저항성 유전자 분석 관련 자료를 추가 제출하도록 규정되었다. 제출된 자료의 검토 결과, 안전성에 문제가 없는 것을 확인 후에 제품에 사용하도록 고시되어 있다. Enterococcus 계열 유산균은 본 연구에서도 확인된 것처럼 단일 유전체 내에 많은 항생제 저항성 유전자를 가지고 있다. 기존 다른 연구에서도 항생제 저항성 유전자의 전파를 매개할 수 있는 저장소(reservoir)와 같은 역할을 하는 것으로 알려져 있다 [14]. 이런 이유로 많은 국가에서 Enterococcus 속의 미생물들은 건강기능식품에서의 사용을 점점 제한하는 추세에 있다. 2021년 식품의약품안전평가원 유산균 안전성 가이드라인에 포함된 항생제 저항성 유전자 관련 내용을 살펴보면 수평적 유전자 이동(horizontal gene transfer) 분자 기작 중에서 plasmid에 의해 진행되는 conjugation과 bacteriophage에 의한 transduction, 그리고 transposon과 같은 mobile element에 의한 전달까지, 총 세 가지에 의해 진행될 수 있는 수평적 유전자 이동을 감안한 항생제 저항성 유전자 분석 가이드를 안내하고 있다. 그러나 가이드에는 항생제 저항성 유전자의 생물정보학적 분석 방법에 대해서는 명확한 기준을 제시하지 않고 있다. 그러나, 본 연구결과에서 확인할 수 있었던 것처럼 분석을 수행하는 tool에 따라서 균주의 항생제 저항성 유전자의 존재 유무가 달라질 수 있는 문제점이 존재한다. 또, 동일서열에 대해 tool마다 다른 판별 알고리즘 및 데이터베이스 사용의 차이에서 결과가 달라지는 문제점 외에도 동일 tool 내에서도 detection parameter의 설정에 따라 항생제 저항성 유전자의 검출 결과가 달라질 수 있는 어려움이 존재한다. 따라서, 해당 가이드에 항생제 저항성 유전자의 생물정보학적 분석과 관련된 보다 자세한 분석법이 필요한 실정이다. 구체적으로는, 현재 사용이 가능한 수많은 분석 tool 중에서 사용 가능한 tool과 parameter 설정에 대한 보다 구체적이고 명확한 분석 기준이 명시되어야 할 것이다.
Enterococcus 에 속하는 고시형 2종의 경우, 본 연구에서 적용된 tool에 관계없이 사용된 322개의 모든 유전체에서 항생제 저항성 유전자가 검출되었다.
Enterococcus 에 포함되는 균주들은 앞서 언급되었던 대로 다른 유산균주에 비해 다수의 항생제 저항성 유전자들을 포함하고 있고 활발한 수평적 유전자 이동을 하는 것으로 알려져 있다. 즉, 장 내에서 항생제 저항성 유전자 전파의 허브와 같은 역할을 수행할 수 있는 가능성이 제기되고 있기 때문에 건강기능식품에 포함되어 다량의 생균으로 사용되는 경우 장내 미생물의 항생제 저항성을 증가시킬 위험성이 존재하는 것이 사실이다. 이런 이유로 Enterococcus 계열의 사전 허가제를 시행하고 있지만 본 연구의 결과에서 알 수 있듯이 Enterococcus계열 유산균의 유전체는 기본적으로 다수의 항생제 저항성 유전자를 가지고 있기 때문에 안전성이 입증된 균주들도 분석법을 변경할 경우 다른 항생제 저항성 유전자를 보유하고 있는 결과가 도출될 수도 있다. 배포된 가이드라인에서는 HGTree[15]와 같은 수평적 유전자 이동 정보를 담고 있는 데이터베이스를 통해 획득성/내재성 유전자를 구별하여 판별하는 것을 안내하고 있다. 그러나 HGTree의 내에 포함된 수평적 유전자 이동의 결과는 예측 결과이며, HGTree의 경우 사용된 대규모 분석의 특성으로 인해 개별 수평적 유전자 이동 이벤트에 대한 통계적 유의성이 검증되지 않는 한계가 있다. 즉, 해당 데이터베이스를 기준으로 획득성/내재성에 대한 유의한 판별은 실질적으로 불가능하다고 볼 수 있다. 또한 가이드라인에 포함되어 있지 않지만 미생물의 수평적 유전자 이동의 또 다른 주요 메커니즘 중의 하나인 transformation의 경우 유전체 내의 존재하는 항생제 저항성 유전자가 mobile element와 관련이 없어도 다른 미생물에 전파될 수 있다. Transformation은 세균의 사멸과정에서 발생된 항생제 저항성 유전자를 포함하는 유전체 절편이 타 균주에 전달되어 수평적 유전자 이동이 진행되는 메커니즘이다. 따라서 유산균의 유전체 상에서 발견되는 항생제 저항성 유전자를 특정 케이스로 나누어 평가하고 허가 여부를 판별하는 것은 항생제 저항성 유전자의 전파를 차단한다는 목적에서 그 기준이 명확하지 않다.
본 연구에서의 결과를 바탕으로 판단했을 때 2종의 Enterococcus 속 유산균은 항생제 저항성과 관련된 분석법에 따라 안전성 평가 결과가 달라질 가능성이 존재한다. 따라서 이런 문제점으로 인한 시장에서 예상되는 혼란을 방지하기 위해서는 앞서 언급된 항생제 저항성 유전자 분석방법의 명확한 표준화와 더불어 Enterococcus 계열 2종에 대해서는 대규모 비교 유전체 분석을 통해 추가적인 가이드를 제시하는 것이 바람직하다고 판단된다. 즉, Enterococcus 계열 동일 종 내에 보편적으로 존재하는 항생제 저항성 유전자와 드물게 존재하여 외부 획득 유전자로 예상되는 항생제 저항성 유전자를 구별하는 분석 가이드가 필요하다. 또한, 실제 병원에서 문제가 되는 주요 항생제 저항성 인자의 범위 설정, 개별 유전자에 대해 서열 유사도에 따라 획득성/내제성을 판단할 수 있는 Enterococcus 유산균 전용 항생제 저항성 유전자 데이터베이스 및 판별 기준을 제시하는 것도 바람직한 방향이다. 결론적으로, 프로바이오틱스로 사용되는 Enterococcus 계열 유산균에 대한 항생제 저항성 유전자 관련 유전자시험법 기준이 기존 가이드라인에 보다 구체적이고 명확하게 구축되어야 할 것으로 판단한다.







